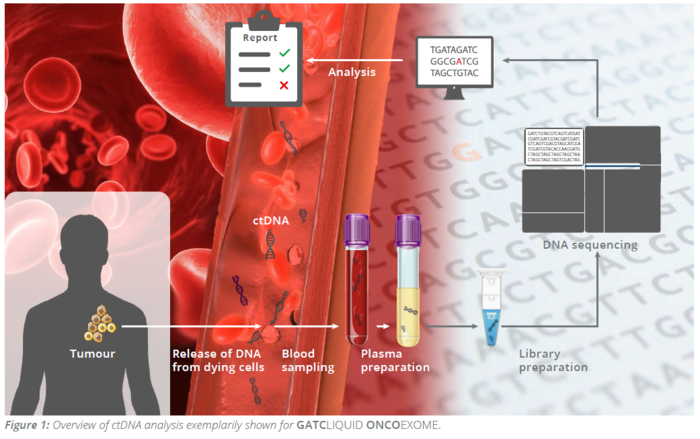
Neinvazivní analýzy cirkulující nádorové DNA (ctDNA) a cirkulujících nádorových buněk (CTC) z krevních vzorků, tzv. tekutá biopsie, v poslední době získávají značnou pozornost v klinické oblasti. Popis cirkulující bezbuněčné DNA není zcela nový a první zmínky lze objevit již v roce 1948, kdy Mandel a Métais popsali extracelulární nukleové kyseliny v krevním oběhu. Avšak až v roce 1970 se vědci začali zajímat o cirkulující bezbuněčnou DNA v souvislosti s nádory a bylo popsáno zvýšené množství DNA v séru pacientů s rakovinou. Přesto, buněčný původ této ctDNA byl nejasný.
V roce 1994 dvě pracovní skupiny nezávisle na sobě publikovaly výsledky týkající se detekce mutací asociovaných s nádorem v DNA z plazmy od pacientů s nádorovým onemocněním, a tak prokázaly, že nádorové buňky mohou uvolňovat DNA do oběhu. Od té doby zájem o detekci a analýzu ctDNA prudce vzrostl a to nejen za účelem detekce nádorů, ale také pro sledování účinnosti terapie a pro včasné zachycení rezistence na léky.
Výzkum ctDNA se dotkl mnoha oblastí klinického výzkumu, včetně:
- transplantačního výzkumu
- detekce virové DNA
- neinvazivního prenatálního testování na chromozomální aberace
Avšak sledování ctDNA skýtá i určitá úskalí jako:
- nízké hladiny cirkulující DNA ve většině případů
- obtížná diskriminace mezi DNA ze zdravých a nádorových buněk
- přesná kvantifikace mutovaných fragmentů přítomných na vysokém pozadí fragmentů divokého typu